Новый метод FI-Chrom создаёт 3D-модели хромосом, раскрывая их динамическую структуру
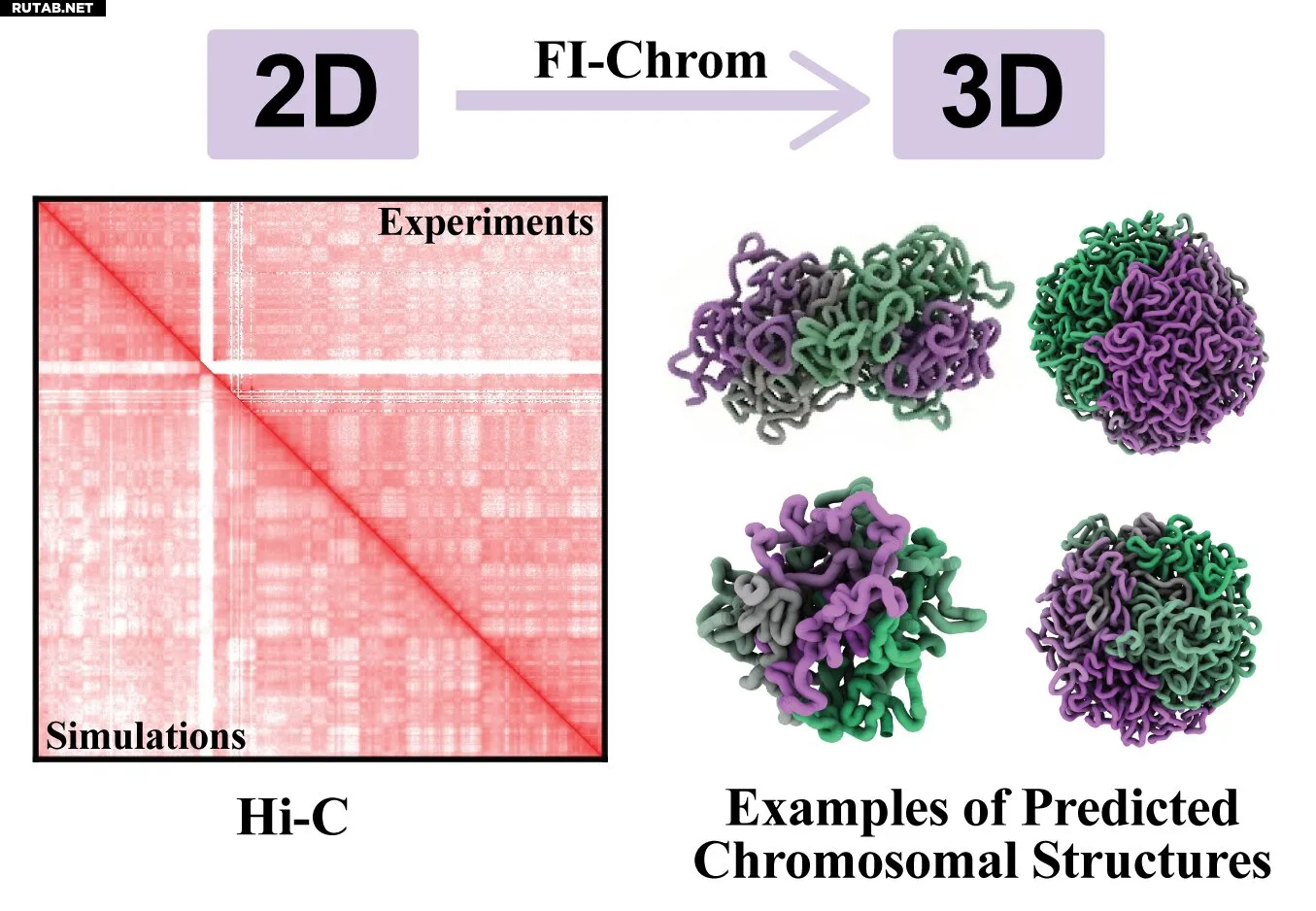
Карта Hi-C (слева) и примеры 3D-моделей белков, сгенерированных на основе карт Hi-C (справа). Автор: Университет Райса
Хромосомы — мастера организации. Эти длинные цепочки ДНК складываются в ансамбль компактных структур, которые держат нужные части генома доступными, одновременно убирая те, что используются реже. Понимание сложности этих структур всегда было сложной задачей; хромосомы — это большие системы, и расшифровка их структуры и динамики требует сочетания экспериментальных данных и теоретических подходов.
Метод FI-Chrom, описанный в недавней публикации Хосе Онучика и Винисиуса Контессото из Университета Райса, представляет собой новый и эффективный подход к созданию 3D-карт хромосом на основе реальных данных.
Исследование опубликовано в журнале Proceedings of the National Academy of Sciences.
FI-Chrom использует данные из карт Hi-C хромосом. Эти карты разбивают хромосому на единицы длины, называемые «бусинами», и показывают, как часто каждая бусина находится близко к другим бусинам. Эта информация показывает только вероятности того, что любые две бусины окажутся соседями, и не содержит прямых трёхмерных данных.
Представьте это как логическую головоломку, где правила или параметры читаются примерно так: «Бусина А с вероятностью 99% находится рядом с бусиной B, с вероятностью 36% — рядом с бусиной C и с вероятностью 62% — рядом с бусиной D». Исследователи знали, что 3D-модель можно построить, разместив каждую бусину в пространстве, не нарушая ни одного из параметров карты Hi-C. Единственная проблема в том, что на картах Hi-C существуют сотни тысяч бусин и десятки миллионов отображённых взаимодействий, показывающих близость бусин.
«У нас были карты хромосом, которые теоретически давали нам 3D-информацию, но мы фактически читали их в 2D-пространстве, — объясняет Онучик, заведующий кафедрой физики имени Гарри Си и Ольги К. Висс и автор-корреспондент исследования. — Теперь мы создали FI-Chrom, программу с открытым исходным кодом, которая может превращать эти карты Hi-C в 3D-модели хромосом».
Антонио Оливейра-младший, научный сотрудник, работающий с Онучиком и Контессото и первый автор статьи, обратился к обратной статистической механике, чтобы решить эту сложную проблему, создав программу FI-Chrom.
«FI-Chrom, доступный для использования любым исследователем, предоставит нам невероятное представление о структуре хромосом у любого организма, от человека до дрожжей и бактерий», — сказал Контессото, доцент-исследователь физики и астрономии.
Хотя Оливейра использовал подход, известный как принцип максимальной энтропии, он не давал FI-Chrom никаких предварительных инструкций о том, как должны выглядеть хромосомы. Вместо этого он обучил программу на экспериментальных данных, пока FI-Chrom не смогла создавать модели, соответствующие известной карте контактов Hi-C.
«Я, например, не говорил программе, что человеческие хромосомы обычно разделяются на два компартмента и минимизируют узлы, — сказал Оливейра. — Тем не менее, после нескольких раундов обучения FI-Chrom начал создавать 3D-модели с этими особенностями. Эти физические особенности нельзя наблюдать непосредственно на картах Hi-C, но они становятся очевидными из предсказанного ансамбля 3D-структур».
FI-Chrom также позволяет исследователям преобразовывать наборы данных Hi-C в динамические механизмы. Хотя данные Hi-C содержат только структурную информацию, исследователи смоделировали различные положения, чтобы вывести движение, поскольку 2D-данные показывают, как часто бусины находятся близко или далеко друг от друга.
«Важно отметить, что 2D-карты Hi-C получаются из популяции клеток и кодируют не единичную структуру хромосомы, а их ансамбль», — сказал Контессото.
«Мы смогли использовать эту динамическую информацию Hi-C для моделирования петель хроматина, — сказал Онучик. — Хотя мы и раньше знали, что петли существуют, с помощью модели FI-Chrom мы смогли продемонстрировать, что петли образуются временно, а не являются статическими особенностями хромосом».
Источник: Rice University






0 комментариев