Учёные создали одну из самых детальных 3D-карт организации хромосом в ядре клетки
В журнале Nature опубликована одна из самых детальных трёхмерных карт, показывающая, как хромосомы человека организованы и упакованы внутри клеточного ядра. Учёные в рамках проекта 4D Nucleome идентифицировали более 140 000 ДНК-петлевых взаимодействий.
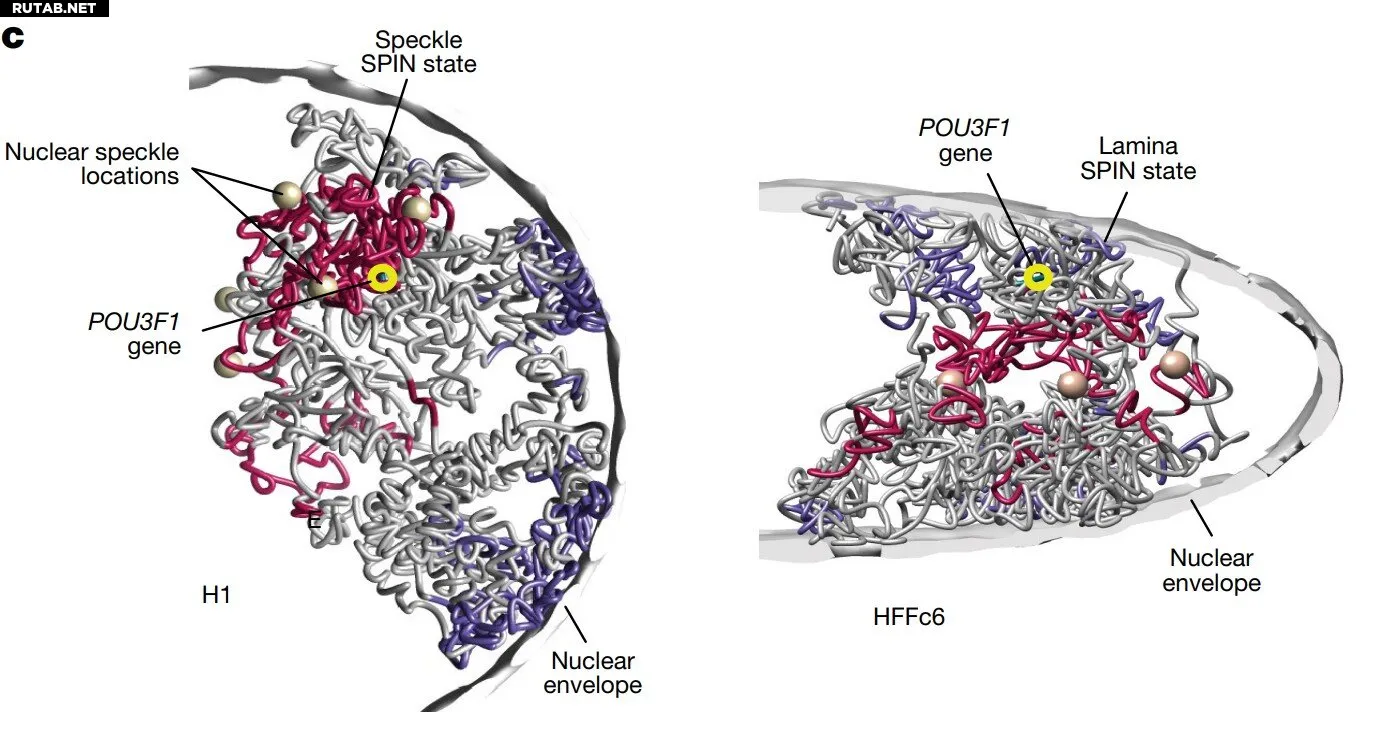
Представительные 3D-структуры хромосомы 1 в клетках H1 (слева) и HFFc6 (справа). Автор: Nature (2025). DOI: 10.1038/s41586-025-09890-3
Хромосомы складываются в специфические трёхмерные формы, а не просто существуют в виде свободных нитей. То, как они сворачиваются, какие структуры образуют и где располагаются, критически важно для правильной работы клетки, экспрессии генов и репликации ДНК.
Целью проекта 4D Nucleome было понять 3D-организацию хромосом в ядре и её изменения во времени. Команда исследовала два типа человеческих клеток: эмбриональные стволовые клетки H1 и фибробласты. Они не только составили карту петель, но и разработали вычислительные методы, способные предсказывать укладку генома только на основе его ДНК-последовательности. Это упрощает определение того, как генетические вариации, в том числе связанные с болезнями, влияют на структуру и функцию генома.
Картирование петель в 3D-пространстве
Проект «Геном человека» в начале XXI века дал первую последовательность генома, но не объяснил, как гены включаются или выключаются. Для этого необходимо понимать архитектуру хромосом и точную карту расположения ДНК в ядре.
В ядре хромосомы организуются в 3D-структуры, где ДНК наматывается на гистоновые белки, а петли формируются с помощью белков когезина. Эти хроматиновые петли, сближая отдельные участки ДНК, играют фундаментальную роль в регуляции генов.
Обзор подхода к генерации и интеграции геномных данных по 4D-нуклеоме. Автор: Nature (2025). DOI: 10.1038/s41586-025-09890-3
Исследователи использовали серию лабораторных тестов (геномных анализов), чтобы измерить частоту взаимодействий между различными участками ДНК. Данные были интегрированы для создания платформы Integrative Genome Modeling (IGM), включающей 1000 различных 3D-моделей генома для отдельных клеток. Эти же данные использовали для обучения моделей глубокого обучения, предсказывающих 3D-укладку генома.
В итоге команда каталогизировала более 141 365 регуляторных петель для стволовых клеток и 146 140 для фибробластов и создала одноклеточные модели, показывающие взаимодействия генов с удалёнными регуляторными элементами в трёхмерном пространстве.
Учёные подчёркивают, что эти модели могут выявить последовательности ДНК, управляющие укладкой генома, и позволить предсказывать генетические вариации, изменяющие эту 3D-структуру. Это открывает новые возможности для исследований генетических заболеваний.
Больше информации: Job Dekker et al, An integrated view of the structure and function of the human 4D nucleome, Nature (2025). DOI: 10.1038/s41586-025-09890-3
Elzo de Wit, Systematic maps reveal how human chromosomes are organized, Nature (2025). DOI: 10.1038/d41586-025-03808-9











0 комментариев