ИИ для предсказания устойчивости к антибиотикам может быть менее точным, чем считалось
Исследователи из Института инфекционных исследований Гельмгольца (HIRI) в Вюрцбурге выяснили, что машинное обучение для прогнозирования антибиотикорезистентности у бактерий часто даёт излишне оптимистичные результаты. Причина — в смещении данных, связанном с эволюционным родством патогенов.
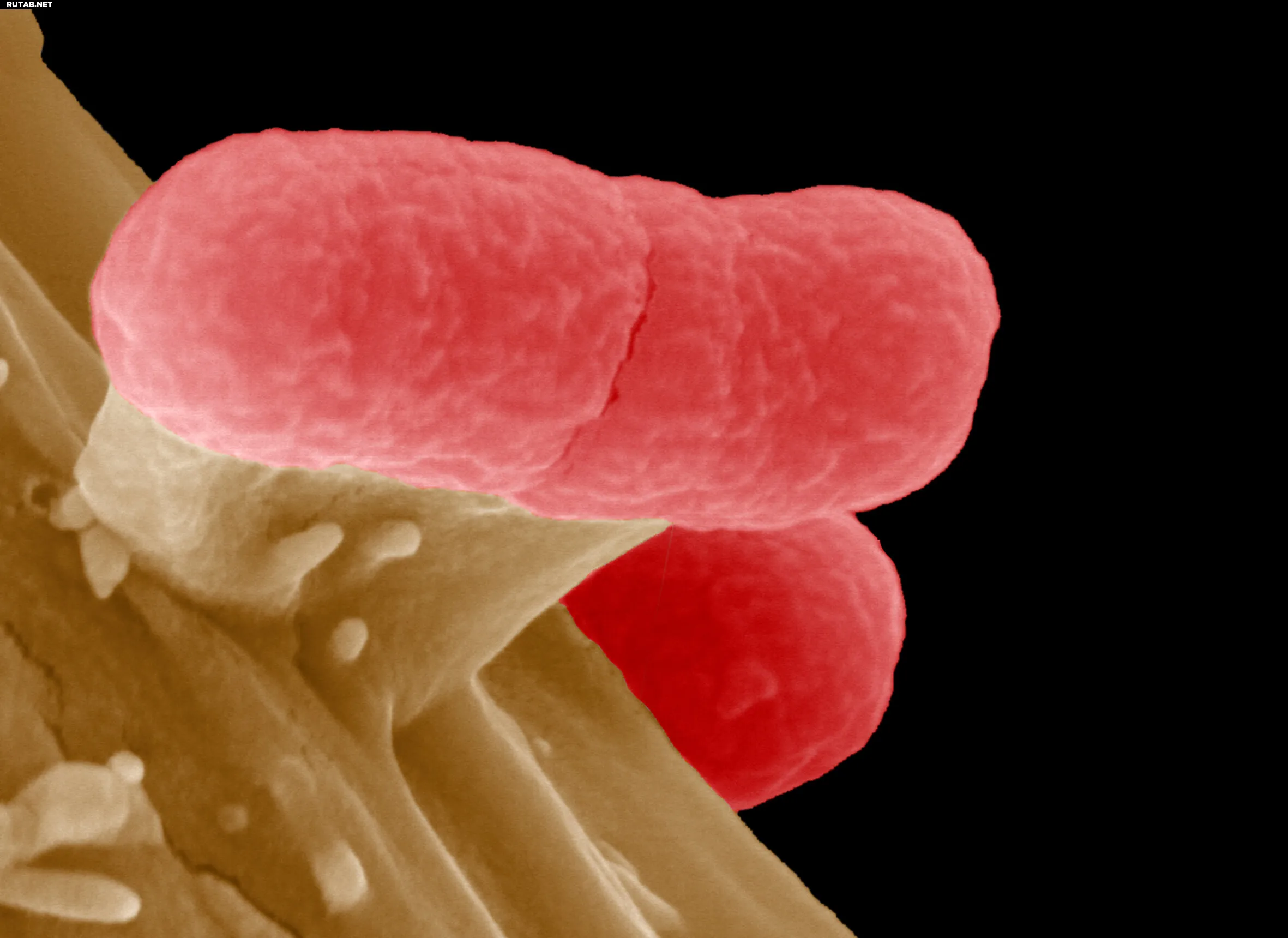
Изображение бактерии кишечной палочки (Escherichia coli) под электронным микроскопом. Автор: Манфред Роде
Алгоритмы обучаются на геномных данных, но если в выборке преобладают близкородственные штаммы, модель может научиться распознавать не сами механизмы устойчивости, а генетические черты, характерные для определённой линии бактерий. Это создаёт ложное впечатление о высокой точности прогноза.
Учёные проанализировали более 24 000 геномов пяти ключевых видов болезнетворных бактерий, включая кишечную палочку (Escherichia coli) и золотистый стафилококк (Staphylococcus aureus). Оказалось, что при более реалистичной оценке, когда обучающие и тестовые данные содержат бактерии из разных генетических семейств, точность моделей существенно падает.
«Когда модели оцениваются более реалистично, с учётом того, что обучающие и тестовые бактерии не происходят из одного генетического семейства, точность снижается — иногда резко», — отмечает первый автор исследования Янъин Ю.
Выводы, опубликованные в журнале PLOS Biology, указывают на необходимость учитывать эволюционную структуру бактериальных популяций при разработке инструментов для прогнозирования резистентности. Это важно для создания надёжных систем, способных давать точные рекомендации по лечению при появлении новых штаммов патогенов.
Больше информации: Yanying Yu et al, Biased sampling driven by bacterial population structure confounds machine learning prediction of antimicrobial resistance, PLOS Biology (2025). DOI: 10.1371/journal.pbio.3003539












0 комментариев