Новый метод позволяет создавать атомные 3D-модели динамики белков
Международная группа исследователей представила вычислительный фреймворк, который позволяет воссоздавать точные трехмерные атомные модели динамических конформаций белков на основе данных высокоскоростной атомно-силовой микроскопии (HS-AFM).
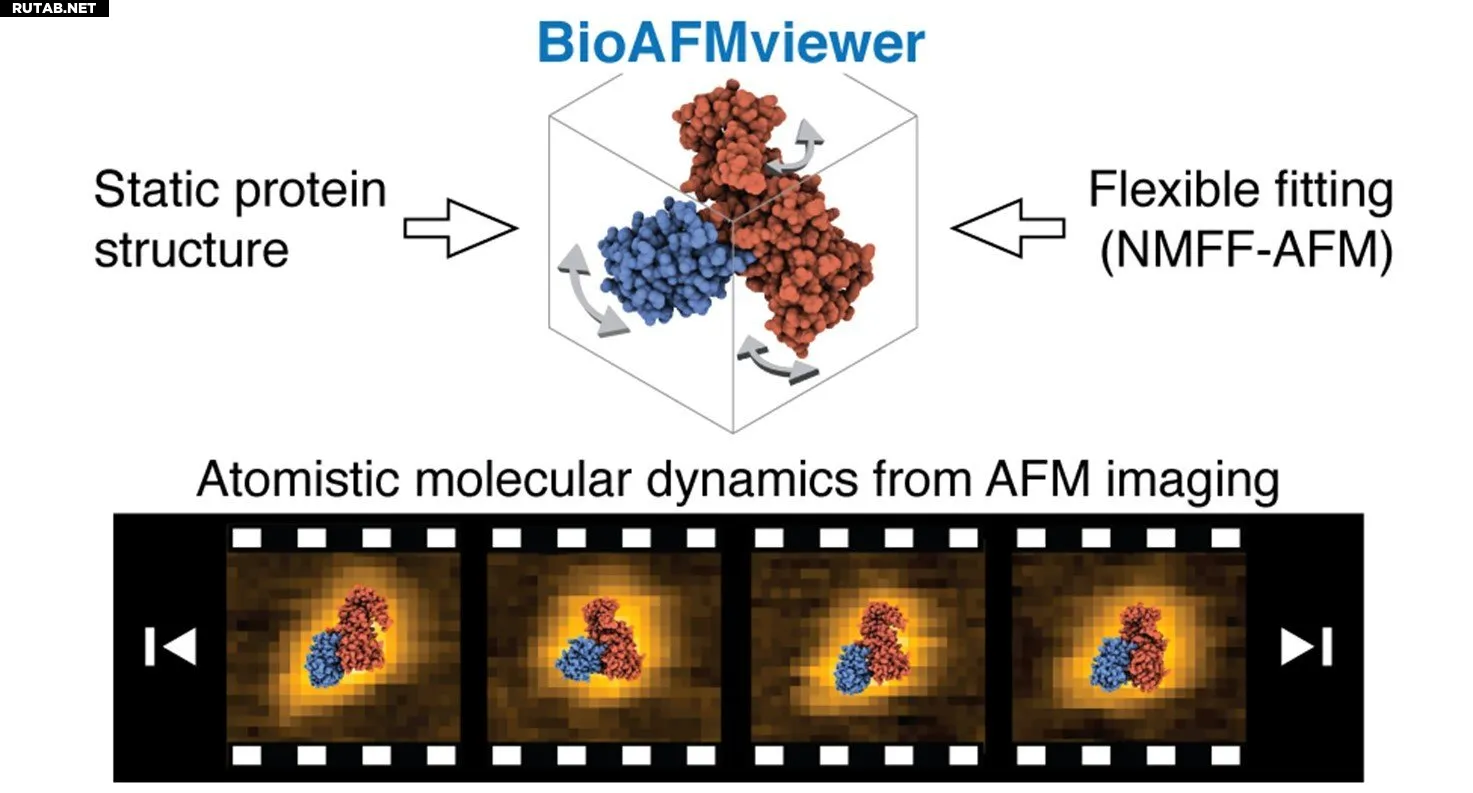
Схема гибкого подгонки BioAFMviewer для определения атомной биомолекулярной динамики из данных HS-AFM. Автор: ACS Nano (2025). DOI: 10.1021/acsnano.5c10073
HS-AFM — единственная экспериментальная методика, позволяющая непосредственно наблюдать белки в динамике. Однако из-за ограниченного пространственного разрешения она не предоставляет достаточной информации для атомарного понимания биомолекулярных функций.
Новый метод гибкого подгонки, разработанный группой Флоренс Тамы и реализованный в программной платформе BioAFMviewer Хольгера Флексига, моделирует конформационную динамику известной статической структуры белка для идентификации атомных моделей, которые наилучшим образом соответствуют экспериментальным AFM-изображениям.
Анализ данных HS-AFM для различных белков показал, что гибкая подгонка может выводить атомные модели, включая движения большой амплитуды, что значительно улучшает понимание функциональной конформационной динамики.
Вычислительная эффективность метода позволяет применять его к крупным белковым сборкам, что авторы продемонстрировали на примере актинового филамента массой 4 мегадальтон, состоящего из около 280 000 атомов. Уникальным достижением стала демонстрация атомного молекулярного фильма о динамике белка, восстановленного из данных HS-AFM.
Программное обеспечение BioAFMviewer доступно для бесплатного скачивания с сайта проекта www.bioafmviewer.com. Исследование опубликовано в журнале ACS Nano.
ИИ: В 2025 году такие разработки открывают новые горизонты в структурной биологии, позволяя буквально "увидеть" атомарную динамику биомолекул, что критически важно для разработки лекарств и понимания фундаментальных биологических процессов.







0 комментариев