ИИ-инструмент предсказывает совместимость ферментов и субстратов
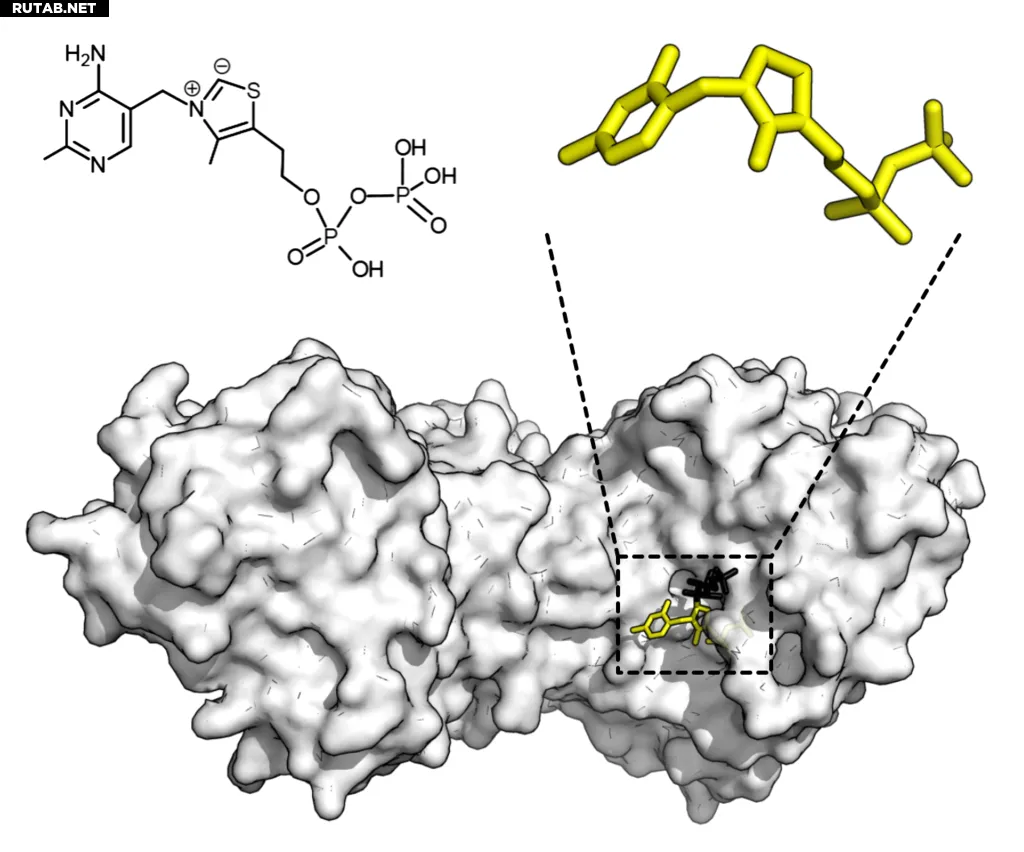
Химическая структура тиаминпирофосфата и белковая структура транскетолазы. Кофактор тиаминпирофосфата показан жёлтым, субстрат ксилулозо-5-фосфат — чёрным. Автор: Thomas Shafee/Wikipedia
Новый инструмент на основе искусственного интеллекта помогает исследователям определять, насколько хорошо фермент сочетается с целевой молекулой. Это позволяет находить оптимальные комбинации ферментов и субстратов для применения в катализе, медицине и промышленности.
Команда под руководством Хуимина Чжао, профессора химической и биомолекулярной инженерии в Университете Иллинойса, разработала систему EZSpecificity, используя новые данные о парах «фермент-субстрат» и специальный алгоритм машинного обучения. Инструмент доступен бесплатно онлайн, а результаты работы опубликованы в журнале Nature.
«Если мы хотим получить определённый продукт с помощью фермента, нам нужна наилучшая комбинация фермента и субстрата», — пояснил Чжао. — «EZSpecificity — это модель ИИ, которая анализирует последовательность фермента и предсказывает, какой субстрат лучше всего подойдёт для него. Она отлично дополняет модель CLEAN, которую мы разработали более двух лет назад для предсказания функции фермента по его последовательности».
Ферменты — это крупные белки, катализирующие молекулярные реакции. Они имеют карманообразные области, в которые встраиваются целевые молекулы, называемые субстратами. Степень соответствия фермента и субстрата называется специфичностью. Классическая аналогия — замок и ключ: только правильный ключ откроет замок. Однако, как отметил Чжао, работа ферментов сложнее.
«Сложность в том, что карман не статичен. Фермент меняет конформацию при взаимодействии с субстратом. Это скорее индуцированное соответствие. Некоторые ферменты многофункциональны и могут катализировать разные типы реакций. Это сильно затрудняет прогнозирование. Поэтому нам и нужна модель машинного обучения и экспериментальные данные, которые действительно докажут, какая пара сработает лучше», — сказал Чжао.
Существующие модели предсказания специфичности ферментов ограничены по точности и типам реакций, которые они могут предсказать.
Группа Чжао поняла, что для улучшения прогнозирования необходимо расширить набор данных для обучения модели. Они сотрудничали с командой Дивакара Шукла, профессора химической и биомолекулярной инженерии. Группа Шукла провела докинговые исследования для различных классов ферментов, создав обширную базу данных, содержащую информацию не только о последовательности и структуре ферментов, но и о том, как ферменты разных классов взаимодействуют с различными субстратами.
«Эксперименты по изучению взаимодействия ферментов с субстратами часто медленные и сложные, поэтому мы провели масштабные докинговые симуляции, чтобы дополнить существующие экспериментальные данные», — сказал Шукла. — «Мы изучили взаимодействия на атомном уровне. Миллионы расчётов докинга дали нам недостающий элемент пазла для создания высокоточного предсказателя специфичности ферментов».
Исследователи протестировали EZSpecificity в четырёх сценариях, имитирующих реальные задачи. Новая модель превзошла текущего лидера — ESP — во всех случаях. Затем учёные экспериментально проверили EZSpecificity на восьми ферментах-галогеназах и 78 субстратах. EZSpecificity показала точность 91,7% для лучших предсказанных пар, в то время как ESP — только 58,3%.
«Я не могу утверждать, что это работает для каждого фермента, но для определённых ферментов EZSpecificity действительно показала отличные результаты», — сказал Чжао. — «Мы хотим, чтобы этим инструментом могли пользоваться другие, поэтому создали пользовательский интерфейс. Теперь исследователи могут ввести субстрат и белковую последовательность, и наш инструмент предскажет, насколько хорошо они будут работать».
В дальнейшем команда планирует расширить инструменты ИИ для анализа селективности ферментов, что поможет исключить ферменты с нецелевыми эффектами, а также продолжит улучшать EZSpecificity, добавляя новые экспериментальные данные.
Дополнительная информация: Haiyang Cui et al, Enzyme specificity prediction using cross attention graph neural networks, Nature (2025). DOI: 10.1038/s41586-025-09697-2











0 комментариев