Ученые создали карту генов бактерий для поиска новых антибиотиков
Исследователи из Медицинской школы Йонг Лоо Лин Национального университета Сингапура и Калифорнийского университета в Беркли разработали новый метод Dual Tn-seq, позволяющий быстро идентифицировать генетические взаимодействия в бактериях. Эта технология выявляет уязвимости, которые могут стать мишенями для антибиотиков следующего поколения.
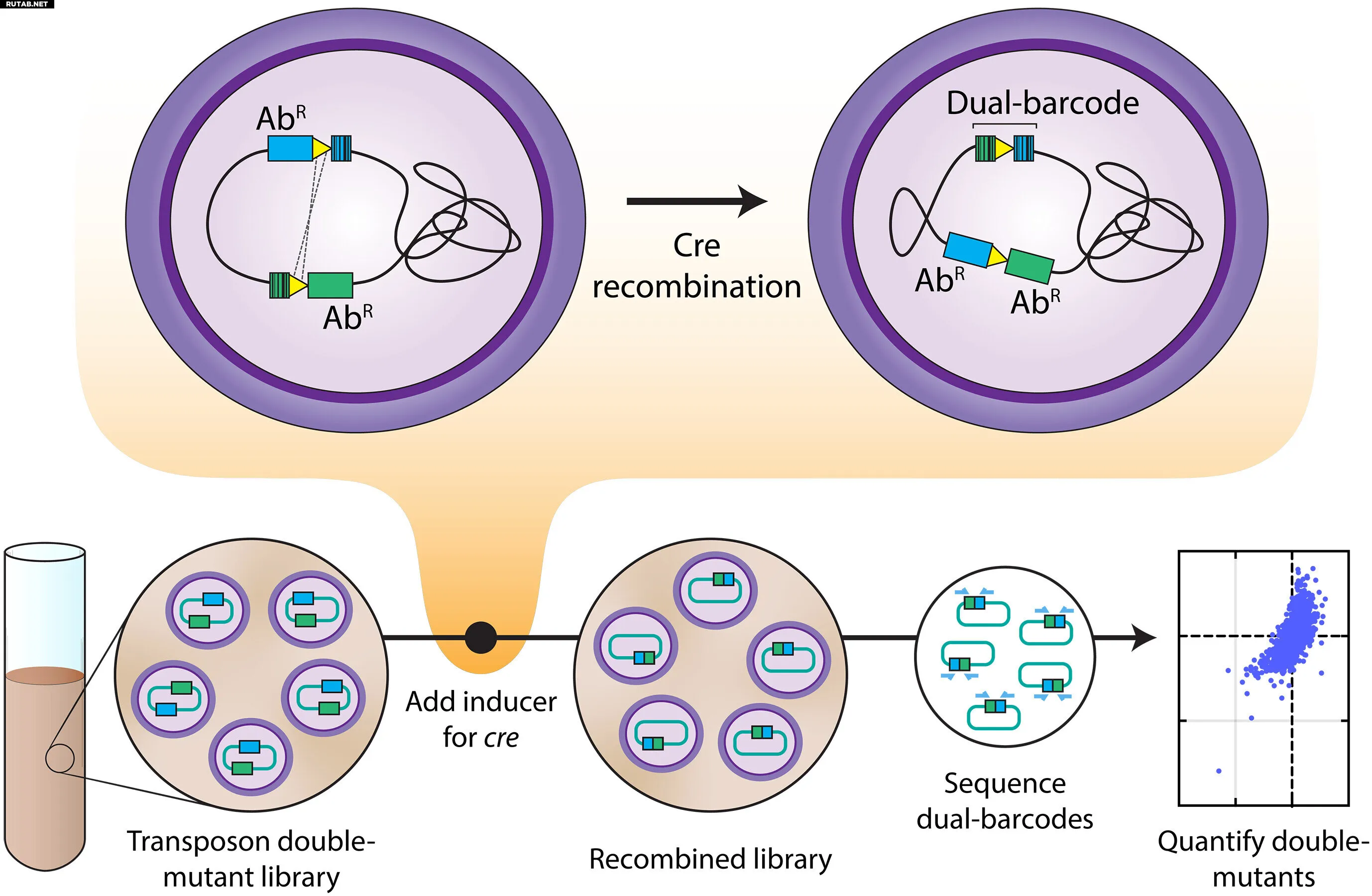
Метод Dual Tn-seq исследует генетические взаимодействия в бактериях. Автор: Science (2025). DOI: 10.1126/science.adt7685
«Это похоже на карту социальной сети для бактериальных генов», — пояснил руководитель исследования доцент Крис Шам Лок То. «Теперь мы можем видеть, какие гены зависят друг от друга, и какие пары генов являются для бактерий критически важными».
Метод использует мобильные генетические элементы — транспозоны, каждый с уникальным ДНК-«штрихкодом». Применяя фермент Cre-рекомбиназу, ученые смогли анализировать двойные мутации в масштабе всего генома. В исследовании на Streptococcus pneumoniae было проанализировано 73% из 1,3 миллиона возможных парных делеций генов.
Команда обнаружила 244 важных генетических партнерства, включая смертельные комбинации, где удаление обоих генов приводило к гибели бактерии. Также был идентифицирован новый фермент PyrJ, производящий строительные блоки ДНК, который присутствует у нескольких патогенных бактерий, включая Clostridioides difficile.
Ученые также определили белок YjbK, который действует как «стартовый выключатель» для построения бактериальной клеточной стенки, и установили возможные функции для 67 ранее неизученных белков.
«Анализируя пары генов, а не по одному, мы можем обнаружить скрытые уязвимости бактерий», — отметил профессор Адам Дойчбауэр из Калифорнийского университета в Беркли.
В перспективе метод планируется усовершенствовать для изучения essential-генов и применить к другим клинически значимым микроорганизмам.










0 комментариев