Учёные обнаружили новые механизмы устойчивости к антибиотикам с помощью следовых количеств ДНК
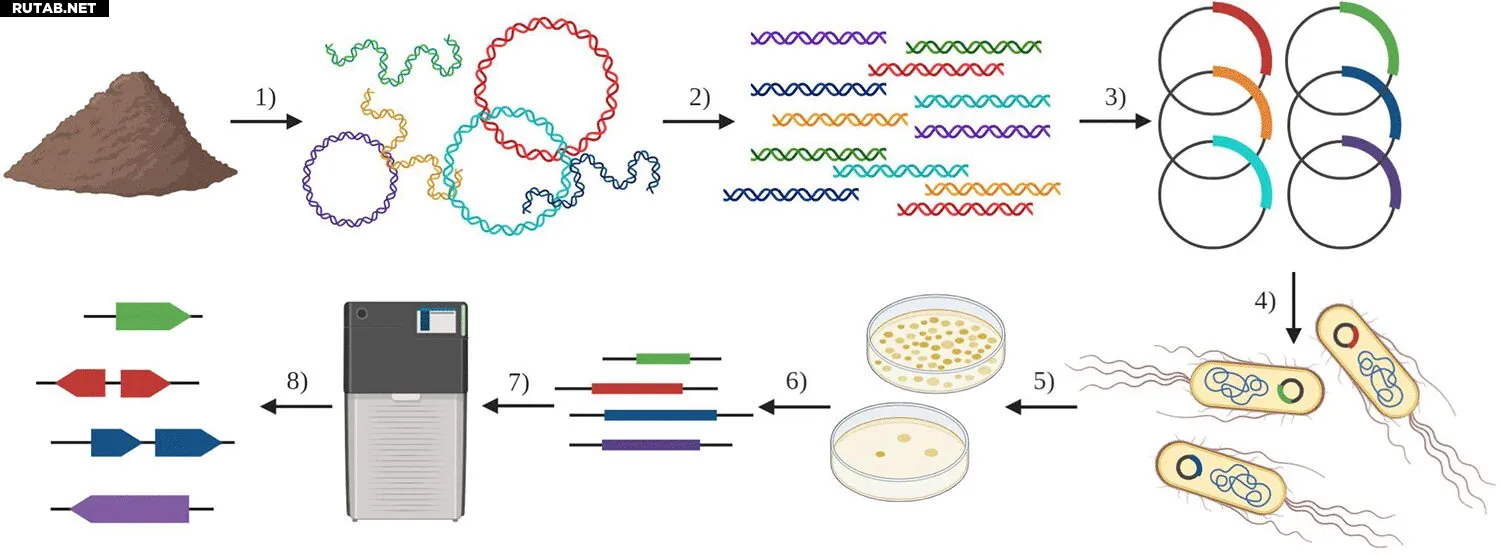
Функциональная метагеномная библиотека. Общий процесс создания и использования функциональных метагеномных библиотек для захвата и обнаружения генов из метагеномов. Автор: mSystems (2021). DOI: 10.1128/mSystems.00524-21
Исследователи из Университета Иллинойса в Урбане-Шампейне разработали метод выделения генов из настолько малых количеств микробной ДНК, что 20 000 образцов понадобилось бы, чтобы их общий вес сравнялся с весом одной крупинки сахара.
В новой статье, опубликованной в mSystems, учёные обнаружили ранее неизвестные гены устойчивости к антибиотикам в бактериальной ДНК, выделенной из человеческого стула и из аквариумов чикагского аквариума Шедда.
«С ростом устойчивости к антибиотикам как никогда важно понимать всё разнообразие механизмов, которые бактерии могут использовать для инактивации или избегания антибиотиков», — сказал Теренс Крофтс, доцент кафедры зоотехнии.
Крофтс разработал метод, известный как METa assembly, чтобы улучшить инструмент микробиологии — функциональную метагеномную библиотеку, которая позволяет исследователям захватывать бактериальные гены из окружающей среды. Метод требует в 100 раз меньше ДНК, чем стандартные функциональные метагеномные библиотеки, что полезно там, где микробы редки или когда исследователи не могут взять большие образцы.
Вместо секвенирования ДНК исследователи используют фермент, чтобы разрезать её на фрагменты размером с ген, которые затем вводят в бактерии E. coli в лаборатории. E. coli, которая легче растёт в лабораторных условиях, чем многие другие микробы, включает чужеродную ДНК в свой генетический аппарат и начинает проявлять её черты.
«Если у E. coli есть ген устойчивости, она может выжить при воздействии антибиотика. Если нет — она погибает», — объяснил Крофтс.
Команда протестировала метод на образце воды из большого аквариума в чикагском аквариуме Шедда, где микробы гораздо менее многочисленны, чем в других средах, таких как почва. Они также протестировали крошечный образец человеческих фекалий.
Исследователи не только создали библиотеки из этих образцов, но и сделали новые открытия о том, как микробы сопротивляются антибиотикам. Например, в тетрациклин-устойчивых последовательностях из аквариума Шедда они идентифицировали новые типы эффлюксных насосов — белковых каналов, которые выводят тетрациклин из клеток.
Интересно, что некоторые колонии E. coli из образца человеческого стула оказались устойчивы к группе антибиотиков, известных как стрептотрицины. Эти антибиотики тестировались в 1940-х годах, но так и не вышли на рынок из-за токсичности для почек у млекопитающих.
«Мы обнаружили то, что выглядит как совершенно новое семейство белков устойчивости к стрептотрицину в наших последовательностях», — сказал Крофтс.
Крофтс планирует применять свой метод METa assembly в сельскохозяйственных системах — отбирая образцы почвы и от скота «от носа до хвоста», поскольку устойчивость к антибиотикам не только возникает на ферме, но часто и начинается там.
ИИ: Это исследование демонстрирует важность поиска новых механизмов устойчивости к антибиотикам в самых неожиданных местах. В 2025 году проблема антибиотикорезистентности остаётся одной из ключевых угроз для глобального здравоохранения, и подобные методы могут помочь опередить эволюцию бактерий.






0 комментариев