Сложные модели глубокого обучения не превосходят простые методы в предсказании генетических изменений
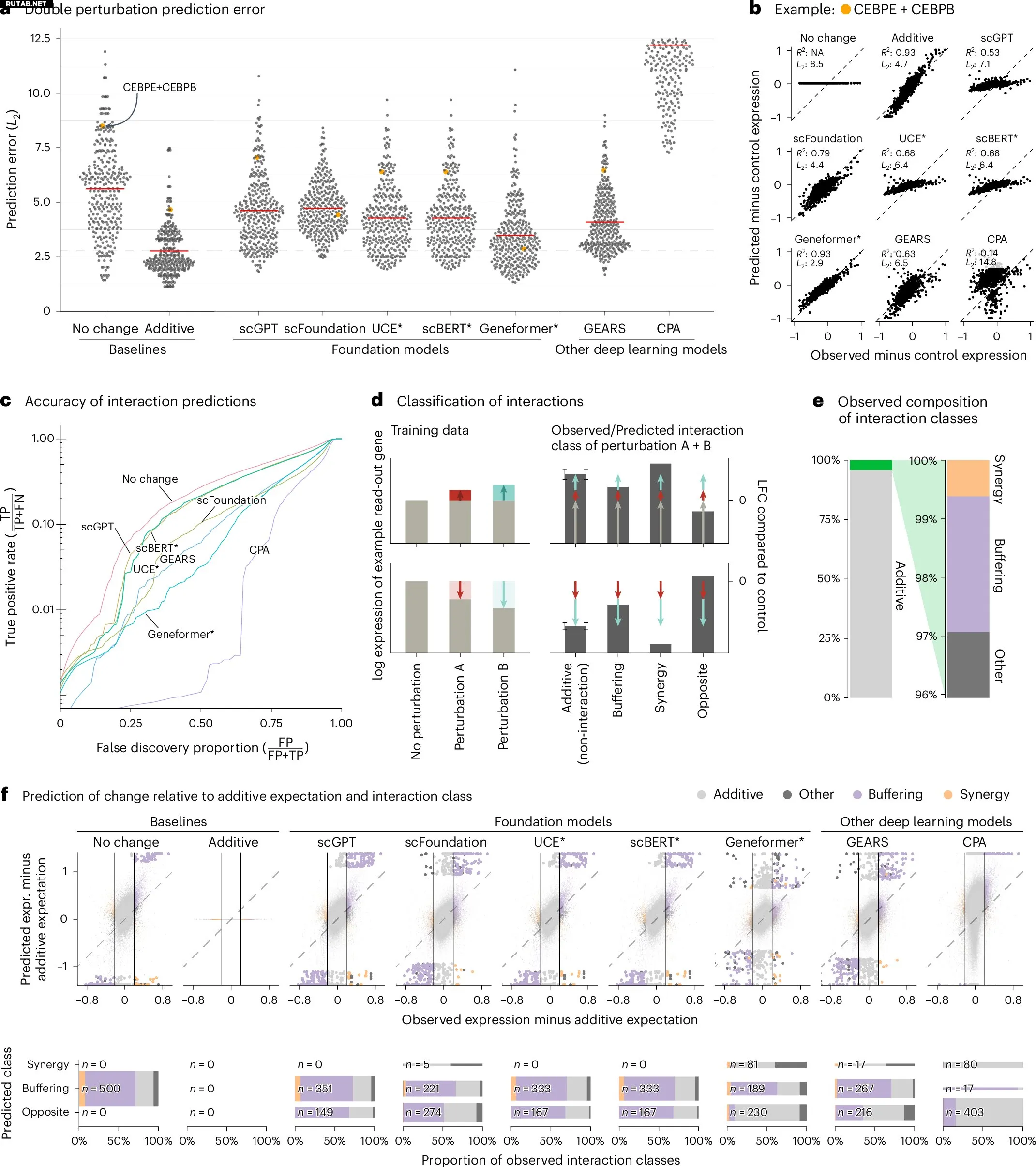
Предсказание двойных генетических изменений. Автор: Nature Methods (2025). DOI: 10.1038/s41592-025-02772-6
Модели глубокого обучения демонстрируют большой потенциал в предсказании и проектировании функциональных ферментов и белков. Но распространяется ли это преимущество на другие области биологии?
Вопреки ожиданиям, недавнее исследование показало, что сложные модели глубокого обучения не превосходят простые базовые методы в предсказании того, как генетические изменения — модификации экспрессии или функции генов — влияют на транскриптом (профиль экспрессии генов в клетках). В случае двойных изменений, когда одновременно модифицируются два гена, ошибка предсказания у сложных моделей оказалась выше, чем у простых аддитивных моделей, которые просто суммируют эффекты изменений генов.
Фундаментальные модели — это модели глубокого обучения, обученные на огромных массивах данных. В данном контексте речь идет о моделях для одноклеточного анализа, обученных на недавно опубликованных транскриптомных данных, охватывающих миллионы клеток.
Исследование, опубликованное в журнале Nature, использовало общедоступные наборы данных CRISPR-изменений в одноклеточных организмах для сравнения пяти ведущих фундаментальных моделей (включая scGPT и scFoundation) и двух других моделей глубокого обучения с четырьмя простыми базовыми методами.
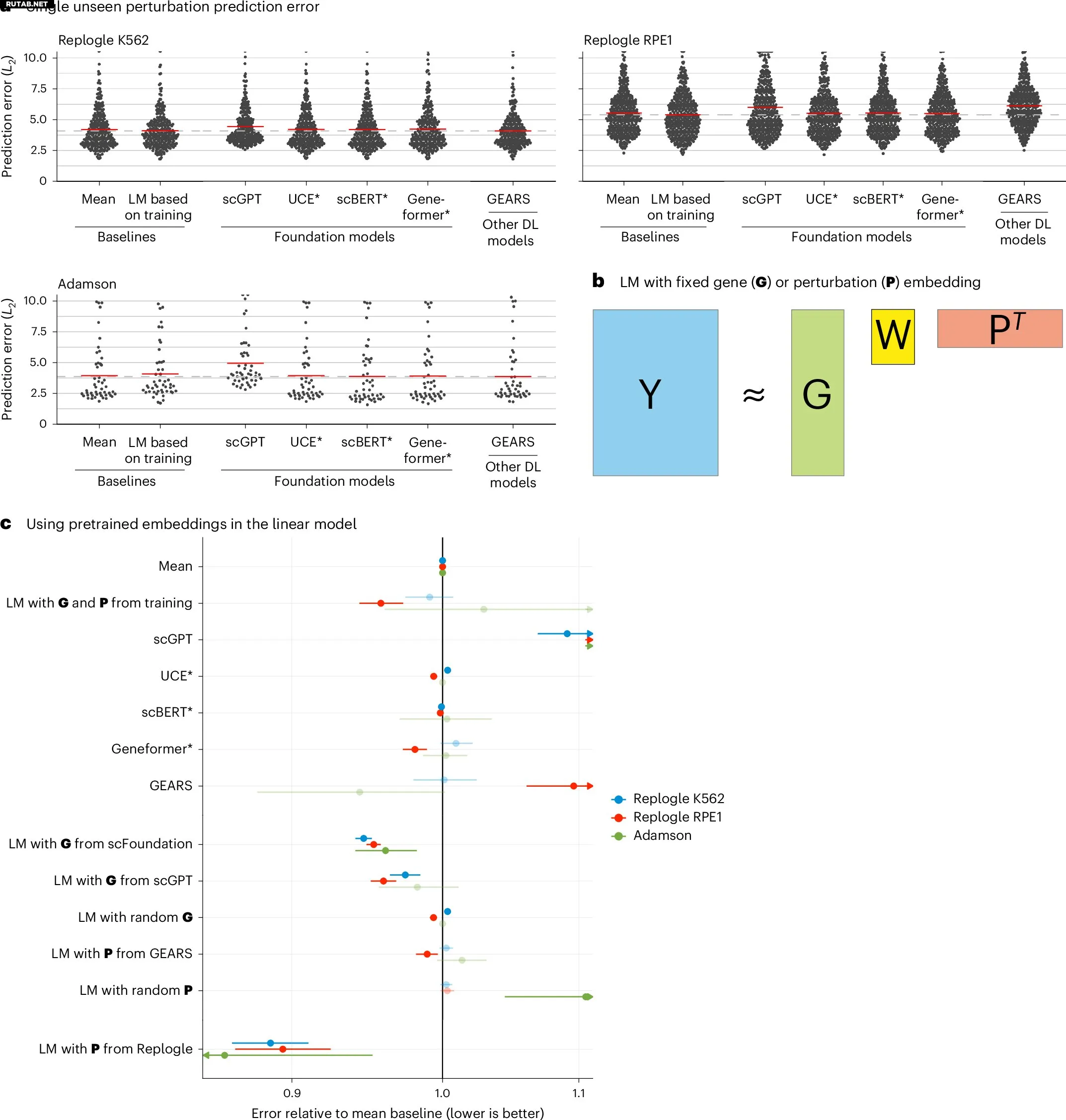
Предсказание единичных генетических изменений. Автор: Nature Methods (2025). DOI: 10.1038/s41592-025-02772-6
Современные исследования фундаментальных моделей глубокого обучения направлены на революцию в понимании биологии за счет обучения на огромных массивах данных. Ожидается, что такие модели смогут получить общее представление о работе клеток, а не просто запоминать результаты конкретных экспериментов. Это позволит предсказывать результаты без проведения экспериментов, что значительно ускорит разработку лекарств и исследования заболеваний.
Однако биология — чрезвычайно сложная наука, где поведение клеток, генов и организмов зависит от множества факторов, многие из которых остаются неизученными. Модели, разрабатываемые для понимания этих сложностей, требуют огромных вычислительных ресурсов — времени, энергии и мощных компьютеров. Прежде чем вкладывать дополнительные ресурсы в создание таких моделей, важно задаться вопросом: действительно ли они эффективнее уже существующих?
Хотя предыдущие исследования проводили сравнительные тесты, большинство из них сравнивало сложные модели между собой, без сопоставления с простыми методами. Авторы данной работы решили исправить это, сравнив простые и интерпретируемые базовые модели со сложными.
Они обнаружили, что ни одна из сложных моделей не превзошла простые базовые методы (такие как предсказание без изменений, среднее значение или линейные модели) в прогнозировании эффекта единичных или двойных генетических изменений на экспрессию генов. Большинство моделей также плохо справлялись с точным предсказанием сложных генетических взаимодействий.
Эти результаты ясно показали, что более высокая стоимость и сложность не обязательно обеспечивают лучшее качество по сравнению с простыми и менее ресурсоемкими методами. Также было подчеркнуто значение строгого тестирования и сравнения новых моделей с существующими.
Исследователи пришли к выводу, что амбициозная цель фундаментальных моделей — получить обобщаемое понимание клеточных состояний и предсказывать результаты на основе этих знаний — пока остается недостижимой.
Дополнительная информация: Constantin Ahlmann-Eltze et al, Deep-learning-based gene perturbation effect prediction does not yet outperform simple linear baselines, Nature Methods (2025). DOI: 10.1038/s41592-025-02772-6











0 комментариев