Ученые создали инструменты для чтения скрытых инструкций ДНК
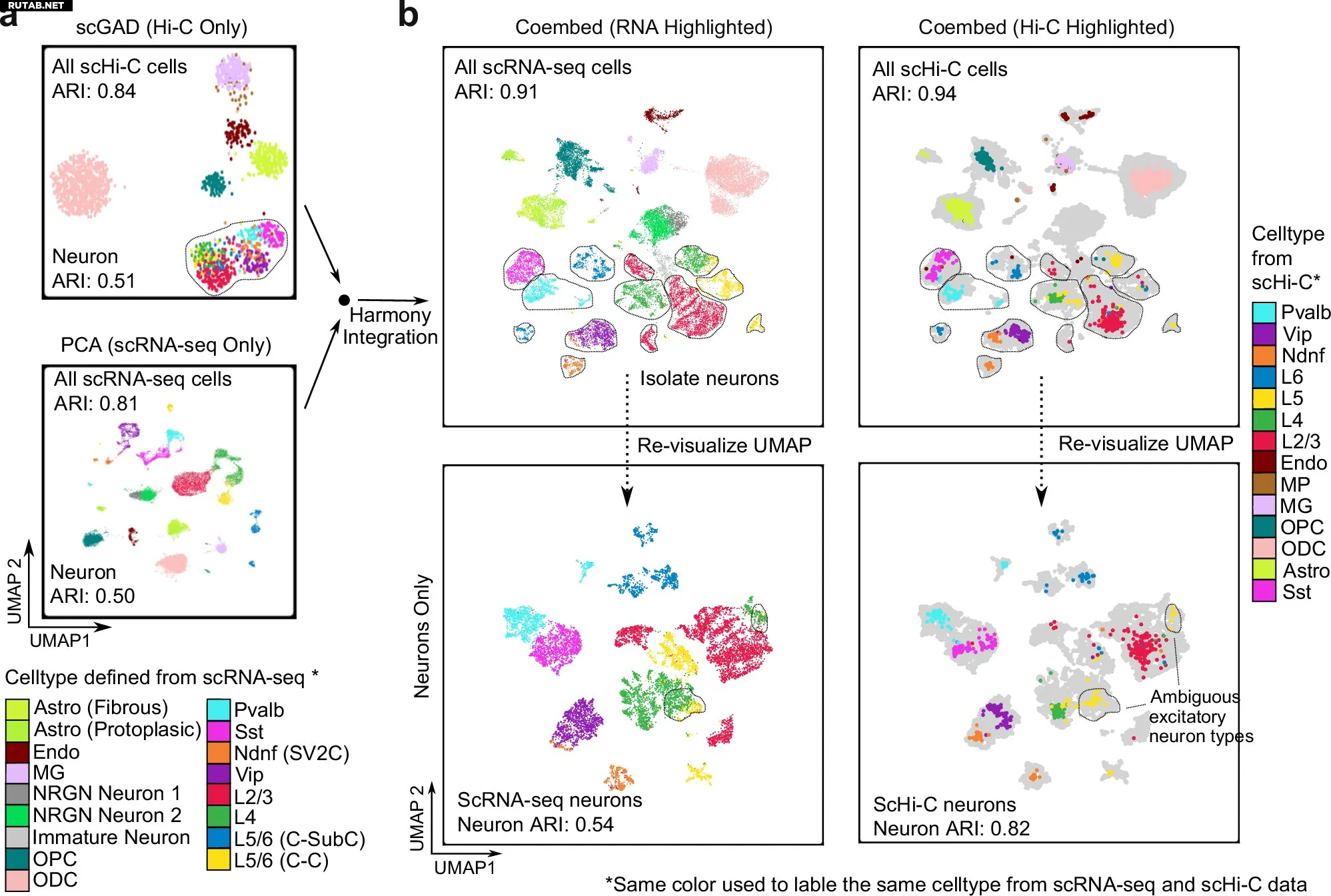
Интеграция с данными scRNA-seq улучшает производительность scHi-C. Автор: Nature Communications (2025). DOI: 10.1038/s41467-025-64186-4
ДНК — это не просто длинная строка генетического кода, а сложная трехмерная структура, свернутая внутри каждой клетки. Это означает, что инструменты для изучения ДНК должны быть столь же сложными — способными читать не только сам код, но и то, как он расположен в пространстве.
Исследователи из Университета Кейс Вестерн Резерв сравнили различные компьютерные инструменты, используемые для анализа того, как ДНК сворачивается и взаимодействует внутри отдельных клеток. Их работа, опубликованная в Nature Communications, может помочь ученым лучше понять, как читать «инструкционное руководство» организма в разных обстоятельствах — например, понимать, что идет не так при развитии болезней или как клетки меняют свои функции по мере нашего роста.
«Трехмерная структура ДНК влияет на то, как гены взаимодействуют друг с другом, подобно тому, как планировка дома влияет на перемещение людей по нему, — объяснил Фулай Джин, профессор кафедры генетики и геномных наук в Медицинской школе Кейс Вестерн Резерв. — Понимание этой структуры имеет решающее значение для выяснения того, как развиваются болезни и как мы можем их лечить».
Команда решала ключевую проблему: существующие инструменты для анализа структуры ДНК часто давали противоречивые результаты. «Это похоже на наличие нескольких переводчиков, которые не могут договориться о том, что означает текст на иностранном языке», — сказал он.
В исследовании вместе с Джином участвовали Цзин Ли, профессор Артура Л. Паркера на кафедре компьютерных и информационных наук в Инженерной школе Кейс, и Янь Ли, доцент и заместитель заведующего кафедрой генетики и геномных наук по научной работе.
Исследователи протестировали 13 программных инструментов на 10 наборах данных от мышей и людей и обнаружили, что разные компьютерные инструменты работают лучше для разных типов данных. Они также выяснили, что изменение способа подготовки данных перед анализом может значительно улучшить результаты. Программы искусственного интеллекта особенно хорошо работают с некачественными и сложными наборами данных.
«По сути, мы помогаем ученым найти или построить лучшие микроскопы, чтобы увидеть, как ДНК работает внутри отдельных клеток, — сказал Джин. — Это может привести к лучшему пониманию генетических заболеваний и потенциально новым стратегиям лечения».
Джин отметил, что улучшенные инструменты могут помочь ученым увидеть, какие гены включаются или выключаются в больных клетках, объяснить, почему лечение работает для одних пациентов, но не для других, и отслеживать, как клетки меняются на ранних стадиях развития.
Исследовательская группа также создала программный пакет, который другие ученые могут использовать для поиска лучшего метода анализа своих конкретных исследований — подобно тому, как приложение GPS находит лучший маршрут до вашего пункта назначения.
«Вместо того чтобы исследователям приходилось гадать, какой инструмент может работать лучше, наше программное обеспечение может протестировать несколько подходов и порекомендовать оптимальный», — сказал Джин.
Методы бесплатно доступны ученым по всему миру через GitHub — платформу с открытым исходным кодом, которая позволяет разработчикам создавать, хранить, управлять и делиться своим кодом. Джин заявил, что такая широкодоступность имеет потенциал для ускорения открытий в различных областях биомедицинских исследований.
«Это значительный шаг к осмыслению массивных генетических данных современного секвенирования — и к пониманию того, как на самом деле работает наш генетический план», — заключил Джин.
Дополнительная информация: Dylan Plummer et al, A comprehensive benchmark of single-cell Hi-C embedding tools, Nature Communications (2025). DOI: 10.1038/s41467-025-64186-4
Источник: Case Western Reserve University









0 комментариев