Новый ИИ-инструмент BioEmu моделирует динамику белков, ускоряя разработку лекарств
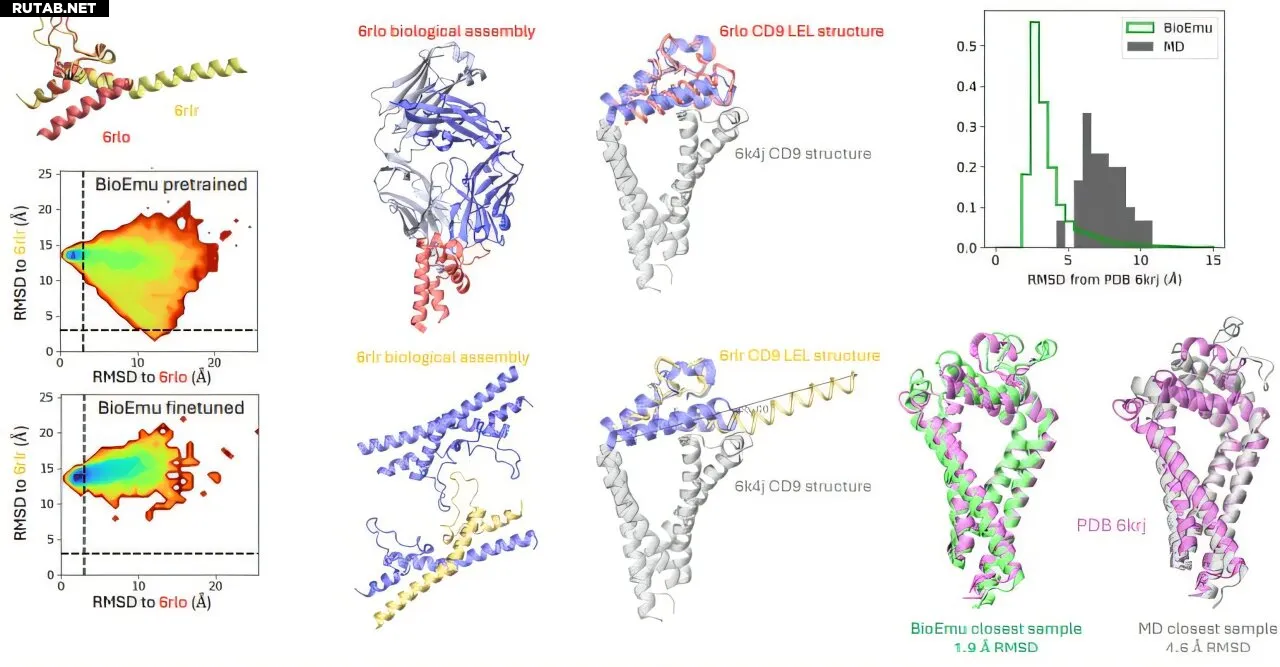
Белок Tetraspanin CD9: сравнение кристаллографических структур, данных BioEmu и молекулярной динамики (MD). Автор: Science (2025). DOI: 10.1126/science.adv9817
Крупный научный прорыв в моделировании белков, разработанный Microsoft Research AI for Science, опубликован в журнале Science. Исследование представляет BioEmu — генеративную систему глубокого обучения, которая с беспрецедентной скоростью и точностью имитирует равновесное поведение белков.
Поскольку биологическая функция белков зависит от динамических изменений их структуры, возможность быстро и точно предсказывать эти изменения открывает новые горизонты для рационального дизайна лекарств, что может снизить процент неудач в клинических испытаниях.
BioEmu способен генерировать тысячи статистически независимых белковых структур в час на одном графическом процессоре (GPU). «Это сокращает стоимость и время, необходимое для анализа функциональных изменений структуры белков», — отмечает профессор Франк Ноэ, руководивший проектом.
Система интегрирует более 200 миллисекунд молекулярно-динамического моделирования с экспериментальными данными, чтобы предсказывать структурные ансамбли и термодинамические свойства с точностью, близкой к экспериментальной.
BioEmu фиксирует сложные биологические явления, такие как формирование скрытых связывающих карманов, движение доменов и локальное разворачивание — всё это критически важно для понимания функций белков и разработки лекарств. Система также предсказывает изменения стабильности белков с точностью, сопоставимой с лабораторными экспериментами.
«Таким образом, BioEmu предоставляет масштабируемый метод для моделирования функций белков на геномном уровне», — добавляет профессор Чечилия Клементи.
Код и модель BioEmu доступны бесплатно по лицензии MIT. Вместе с публикацией Microsoft Research также выпустила набор данных молекулярно-динамического моделирования, использованный для обучения системы. Этот набор включает более 100 миллисекунд симуляций для тысяч белковых систем и является крупнейшим общедоступным набором такого рода.
Хотя исследование полностью проводилось в Microsoft, Свободный университет Берлина отмечает вклад своих сотрудников. Проектом руководил Франк Ноэ, партнёр-менеджер Microsoft Research AI for Science в Берлине, который также является почётным профессором Свободного университета. Ключевой вклад внесла Чечилия Клементи, профессор теоретической и вычислительной биофизики, работавшая в Microsoft в качестве приглашённого исследователя.
Дополнительная информация: Sarah Lewis et al, Scalable emulation of protein equilibrium ensembles with generative deep learning, Science (2025). DOI: 10.1126/science.adv9817







0 комментариев