ИИ разрабатывает оптимальные лекарства от рака без предварительных данных
Автор: Адаптировано из Advanced Science (2025). DOI: 10.1002/advs.202502702
Традиционные методы разработки лекарств включают поиск целевого белка (например, рецептора раковой клетки), вызывающего заболевание, и последующий перебор бесчисленных молекулярных кандидатов (потенциальных препаратов), способных связываться с этим белком и блокировать его функцию. Этот процесс дорогостоящий, трудоёмкий и имеет низкий процент успеха.
Исследователи из KAIST разработали модель искусственного интеллекта, которая, используя только информацию о целевом белке, может проектировать оптимальные лекарственные кандидаты без каких-либо предварительных молекулярных данных — открывая новые возможности для фармацевтики. Исследование опубликовано в журнале Advanced Science.
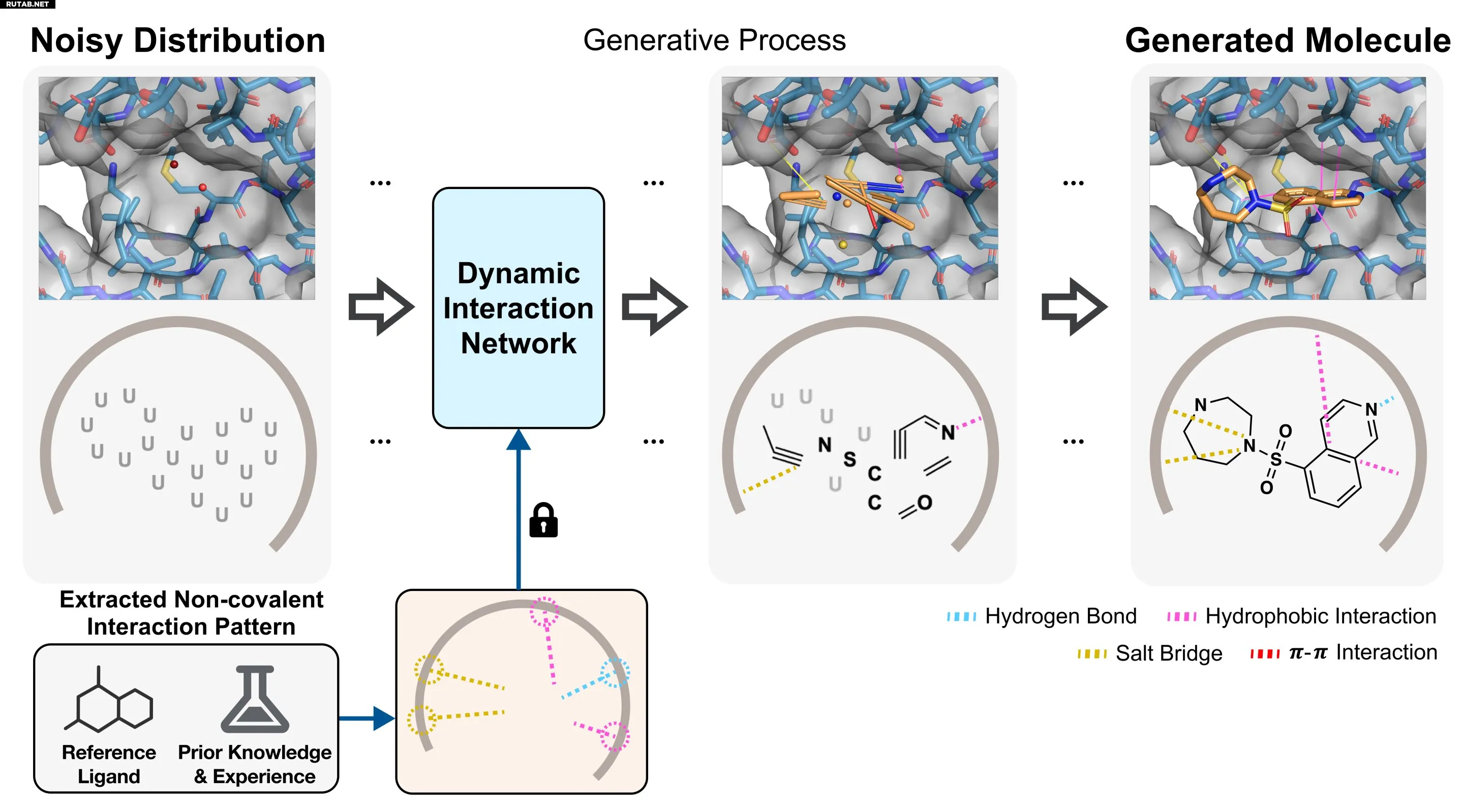
Команда под руководством профессора Ву Ён Кима с кафедры химии создала ИИ-модель под названием BInD (Bond and Interaction-generating Diffusion model). Она способна проектировать и оптимизировать молекулы-кандидаты, ориентируясь исключительно на структуру белка — без необходимости в данных о связывающих молекулах. Модель также предсказывает механизм связывания (нековалентные взаимодействия) между препаратом и целевым белком.
Ключевое отличие этой технологии — подход «одновременного проектирования». Предыдущие ИИ-модели либо генерировали молекулы, либо отдельно оценивали их способность связываться с белком. Новая же модель учитывает механизм связывания уже в процессе генерации, позволяя проводить комплексное проектирование за один шаг.
Поскольку система заранее учитывает критические факторы связывания белка и лиганда, вероятность создания эффективных и стабильных молекул значительно выше. Процесс генерации наглядно демонстрирует, как типы и позиции атомов, ковалентные связи и взаимодействия создаются одновременно, чтобы соответствовать сайту связывания белка.
Кроме того, модель разработана для одновременного соответствия нескольким ключевым критериям: сродство к мишени, «лекарственные» свойства и структурная стабильность. Традиционные системы часто жертвовали одними параметрами ради других, но новая модель балансирует различные цели, повышая практическую применимость.
Исследователи пояснили, что ИИ работает на основе «диффузионной модели» — генеративного подхода, при котором структура постепенно уточняется из случайного состояния. Этот же тип модели используется в AlphaFold 3 — инструменте для генерации структур белок-лиганд, получившем Нобелевскую премию по химии в 2024 году.
В отличие от AlphaFold 3, который предоставляет пространственные координаты атомов, в данном исследовании использовано руководство на основе химических законов — таких как длина связей и расстояния между белком и лигандом. Это позволяет создавать более реалистичные с химической точки зрения структуры.
Команда также применила стратегию оптимизации, при которой успешные паттерны связывания из предыдущих результатов используются повторно. Это позволило модели генерировать ещё более эффективные кандидаты без дополнительного обучения. В частности, ИИ успешно создал молекулы, избирательно связывающиеся с мутированными остатками EGFR — белка, связанного с раком.
Профессор Ву Ён Ким отметил: «Новый ИИ способен изучать и понимать ключевые особенности, необходимые для прочного связывания с целевым белком, и проектировать оптимальные молекулы-кандидаты — даже без каких-либо входных данных. Это может значительно изменить парадигму разработки лекарств».
«Поскольку технология генерирует молекулярные структуры на основе принципов химических взаимодействий, она позволит ускорить и повысить надёжность фармацевтических разработок», — добавил он.
Дополнительная информация: Joongwon Lee et al, BInD: Bond and Interaction-Generating Diffusion Model for Multi-Objective Structure-Based Drug Design, Advanced Science (2025). DOI: 10.1002/advs.202502702
Источник: The Korea Advanced Institute of Science and Technology (KAIST)





0 комментариев